本笔记记录基于R语言的普及性最广、最实用、最具有代表性的5种聚类算法:
-K-均值聚类(K-Means)
-K-中心点聚类(K-Medoids)
-系谱聚类(Hierarchical Clustering, HC)
-密度聚类(Densit-based Spatial Clustering of Application with Noise, DBSCAN)
-期望最大化聚类(Expectation Maximization, EM)
在R中的实现方式如下图所示: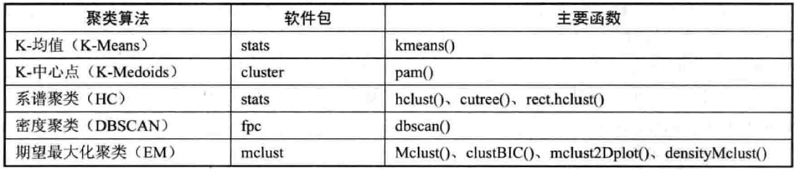
1 数据准备
该数据集含有68个国家和地区的出生率(%)与死亡率(%)。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21> countries = read.csv('countries.csv', head = F)
> head(countries)
V1 V2 V3
1 ALGERIA 36.4 14.6
2 CONGO 37.3 8.0
3 EGYPT 42.1 15.3
4 GHANA 55.8 25.6
5 IVORY COAST 56.1 33.1
6 MALAGASY 41.8 15.8
> names(countries) = c("country", "birth", "death")
> var = countries$country
> var = as.character(var)
> for(i in 1:68) row.names(countries)[i] = var[i]
> head(countries)
country birth death
ALGERIA ALGERIA 36.4 14.6
CONGO CONGO 37.3 8.0
EGYPT EGYPT 42.1 15.3
GHANA GHANA 55.8 25.6
IVORY COAST IVORY COAST 56.1 33.1
MALAGASY MALAGASY 41.8 15.8
接下来选取几个样本点对数据集进行可视化:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21#这部分代码最好在脚本完成,别问我为什么知道
> plot(countries$birth, countries$death)
#获取6个国家和地区在数据集中的位置
> C = which(countries$country == "CHINA")
> T = which(countries$country == "TAIWAN")
> H = which(countries$country == "HONG KONG")
> I = which(countries$country == "INDIA")
> U = which(countries$country == "UNITED STATES")
> J = which(countries$country == "JAPAN")
#获取出生率最高的国家在数据集中的位置
> M = which.max(countries$birth)
#以实心圆点标出如上国家和地区的样本点
> points(countries[c(C,T,H,I,U,J,M),-1], pch = 16)
#标出6个国家和地区样本点的图例
> legend(countries$birth[C]-1.2, countries$death[C], "CHINA", bty="n", xjust=0, yjust=0.5, cex=0.8)
> legend(countries$birth[T], countries$death[T], "TAIWAN", bty="n", xjust=0.5, cex=0.8)
> legend(countries$birth[H], countries$death[H], "HONG KONG", bty="n", xjust=0.5, cex=0.8)
> legend(countries$birth[I]-1.2,countries$death[I], "INDIA", bty="n", xjust=0, yjust=0.5, cex=0.8)
> legend(countries$birth[U], countries$death[U], "UNITED STATES", bty="n", xjust=0.5, yjust=0, cex=0.8)
> legend(countries$birth[J], countries$death[J], "JAPAN", bty="n", xjust=1, yjust=0.5, cex=0.8)
> legend(countries$birth[M], countries$death[M], countries$country[M], bty="n", xjust=1, cex=0.8)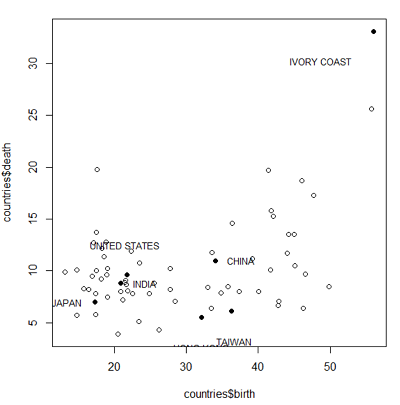
2 聚类分析应用
2.1 K-均值聚类
K-均值聚类是最早出现的聚类分析算法之一,它是一种快速聚类方法,但对于异常值或极值敏感,稳定性差,因此较适合处理分布集中的大样本数据集(可参考Python部分的k-means算法解释)。
调用stats包的kmeans()函数实现:kmeans(x, centers, iter.max=10, nstart=l , algorithm=c("Hartigan-Wong", "Lloyd", "For-gy", "MacQueen"))
在该数据集中,由于事先不知道应该把数据分为几类,因此可通过调节类别参数center的取值,通过组间平方和占总平方和的百分比(即“聚类优度”)来比较选择出最优类别数。由于共有68个样本,可将类别数从1到67取遍:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15> library(stats)
#设置result变量用于存放67个聚类优度值
> result = rep(0, 67)
#对类别数K取1到67进行循环
> for (k in 1:67) {
+ fit_km = kmeans(countries[, -1], centers = k)
+ result[k] = fit_km$betweenss / fit_km$totss
+ }
#输出计算所得resu1t ,取小数位后两位的结果
> round(result, 2)
[1] 0.00 0.72 0.82 0.85 0.86 0.92 0.93 0.93 0.95 0.96 0.96 0.96 0.95 0.97 0.97
[16] 0.98 0.98 0.98 0.99 0.98 0.97 0.98 0.99 0.98 0.99 0.99 0.99 0.99 0.99 0.99
[31] 0.99 0.99 0.99 0.99 0.99 1.00 0.99 0.99 0.99 1.00 0.99 0.99 1.00 1.00 1.00
[46] 1.00 0.99 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00
[61] 1.00 1.00 1.00 1.00 1.00 1.00 1.00
由以上结果大概看出,当类别数约小于10时,随着类别数的增加聚类效果越来越好,但当类别数超过10以后再增加时,聚类效果基本不再提高。
下面对result进行可视化来直观比较各类别数下的聚类优度:1
2
3
4
5> plot(1:67, result, type='b', main='Choosing the Optimal Number of Cluster', xlab='number of cluster:1 to 67', ylab='brtweenss/totss')
#将类别数为10 的点用实心圆标出
> points(10, result[10], pch=16)
#对类别数为10的点给出其坐标标注(x,y),x为其类别数10,y为其聚类优度(%)
> legend(10, result[10], paste('(10,',sprintf('%.1f%%',result[10]*100),')',sep=''), bty='n', xjust=0.3, cex=0.8)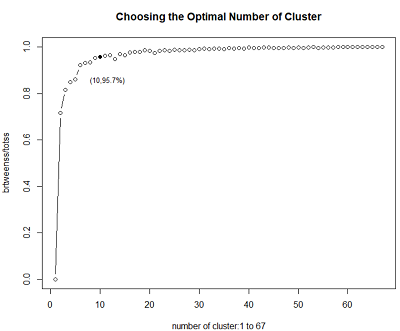
实际上,最优类别数可以是10,也可以是9、11、12等,并无太大差别。在实际选择过程中,如果并非要求极高的聚类效果,取k=5或6即可,较小的类别数在后续的数据分析过程中往往更为方便有效。
接下来对类别数K取10时的聚类结果进行输出:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57> fit_km1 = kmeans(countries[, -1], centers = 10)
> print(fit_km1)
K-means clustering with 10 clusters of sizes 5, 9, 2, 1, 7, 7, 6, 9, 11, 11
Cluster means:
birth death
1 18.32000 6.400000
2 18.17778 11.311111
3 55.95000 29.350000
4 17.60000 19.800000
5 26.24286 7.357143
6 44.04286 16.257143
7 15.28333 8.616667
8 44.37778 8.744444
9 21.75455 8.990909
10 35.09091 9.036364
Clustering vector:
ALGERIA CONGO EGYPT GHANA IVORY COAST
10 10 6 3 3
MALAGASY MOROCCO TUNISIA CAMBODIA CEYLON
6 6 8 6 10
CHINA TAIWAN HONG KONG INDIA INDONESIA
10 10 10 9 5
IRAQ JAPAN JORDAN KOREA MALAYSIA
1 1 8 7 10
MONGOLIA PHILLLIPINES SYRIA THAILAND VIETNAM
10 5 5 10 5
CANADA COSTA RICA DOMINICANR GUATEMALA HONDURAS
5 8 10 6 8
MEXICO NICARAGUA PANAMA UNITED STATES ARGENTINA
8 8 8 9 9
BOLIVIA BRAZIL CHILE COLOMBIA ECUADOR
1 6 10 8 6
PERU URUGUAY VENEZUELA AUSTRIA BElGIUM
5 9 8 2 2
BRITAIN BULGARIA CZECHOSLOVAKIA DENMARK FINLAND
2 7 7 4 2
E.GERMANY W.GERMANY GREECE HUNGARY IRELAND
2 2 1 7 9
ITALY NETHERLANDS NORWAY POLAND PORTUGAL
2 9 2 1 9
ROMANIA SPAIN SWEDEN SWITZERLAND U.S.S.R.
7 9 7 2 9
YUGOSLAVIA AUSTRALIA NEW ZEALAND
9 9 5
Within cluster sum of squares by cluster:
[1] 18.08800 24.76444 28.17000 0.00000 45.19429 69.57429 22.67667
[8] 97.49778 24.53636 123.05455
(between_SS / total_SS = 95.8 %)
Available components:
[1] "cluster" "centers" "totss" "withinss"
[5] "tot.withinss" "betweenss" "size" "iter"
[9] "ifault"
如上结果中显示了10个类别数所含的样本数、各类别中心点坐标、聚类向量(各个国家地区具体属于哪个类别)、以及聚类优度,即组间平方和占总平方和的95.8%,该值可用于与类别数取不同值时的聚类结果进行比较,从而找出最优聚类结果,该百分数越大表明组内差距越小、组间差距越大,即聚类效果越好。
最后简单查看k=10时,与中国大陆属于同一类别的国家和地区有哪些:1
2
3
4
5
6> cluster_CHINA = fit_km1$cluster[which(countries$country == 'CHINA')]
> which(fit_km1$cluster == cluster_CHINA)
ALGERIA CONGO CEYLON CHINA TAIWAN HONG KONG MALAYSIA
1 2 10 11 12 13 20
MONGOLIA THAILAND DOMINICANR CHILE
21 24 28 38
2.2 K-中心点聚类
与K-均值算法在原理上十分相近,针对于K-均值算法易受极值影响这一缺点进行改进。原理上的差异在于选择各类别中心点时不取样本均值点,而在类别内选取到其余样本距离之和最小的样本为中心。
调用Cluster包的pam()函数实现:pam(x, k, diss = inherits(x, "dist") , metric = "euclidean" , medoids = NULL, stand = FALSE, cluster.only = FALSE, do.swap = TRUE , keep.diss = !diss && !cluster.only && n<l00 , keep.data = !diss && ! cluster.only,pamonce = FALSE , trace.lev = 0)
接着上面的案例,看看用K-中心点聚类输出的结果有何不同:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29> library(cluster)
> fit_pam = pam(countries[, -1], 10)
> print(fit_pam)
Medoids:
ID birth death
MALAGASY 6 41.8 15.8
THAILAND 24 34.8 7.9
IVORY COAST 5 56.1 33.1
MOROCCO 7 46.1 18.7
COLOMBIA 39 44.0 11.7
AUSTRALIA 67 21.6 8.7
PERU 41 27.7 8.2
BULGARIA 47 16.4 8.2
NICARAGUA 32 42.9 7.1
BRITAIN 46 18.2 12.2
Clustering vector:
ALGERIA CONGO EGYPT GHANA IVORY COAST
1 2 1 3 3
……
YUGOSLAVIA AUSTRALIA NEW ZEALAND
6 6 7
Objective function:
build swap
2.216542 2.134175
Available components:
[1] "medoids" "id.med" "clustering" "objective" "isolation"
[6] "clusinfo" "silinfo" "diss" "call" "data"
在输出结果中,相对于kmeans()多出了中心点(Medoids)一项,该项指明了聚类完成时各类别的中心点分别是哪几个样本点,它们的变量取值为多少。此外,目标方程项(Objective function) 给出了build 和swap 两个过程中目标方程的值。其中,build过程用于在未指定初始中心点情况下,对于最优初始中心点的寻找;而swap过程则用于在初始中心点的基础上,对目标方程寻找使其能达到局部最优的类别划分状态,即其他划分方式都会使得目标方程的取值低于该值(老实说我不是很理解这个==)。
2.3 系谱聚类
相对于K-均值算法与K-中心点算法,系谱算法的突出特点在于,不需事先设定类别数k,这是因为它每次选代过程仅将距离最近的两个样本/簇聚为一类,其运作过程将自然得到k=1至k=n(n为待分类样本总数)个类别的聚类结果。
调用stats包的hclust()、cutree()、rect.hclust()函数实现:hclust(d, method = "complete", members = NULL)
cutree()函数则可以对hclust()函数的聚类结果进行剪枝,即选择输出指定类别数的系谱聚
类结果。其格式为:cutree(tree, k = NULL, h = NULL)
函数rect.Bclust()可以在plot()形成的系谱图中将指定类别中的样本分支用方框表示出来,十分有助于直观分析聚类结果。其基本格式为:rect.hclust(tree, k = NULL, which = NULL, X = NULL, h = NULL, border = 2, cluster = NULL)
对该案例进行系谱聚类,首先使用dist()函数中默认的欧式距离来生成Countries数据集的距离矩阵,再使用hclust()函数展开系谱聚类,并生成系谱图:1
2
3
4
5
6
7
8
9
10> fit_hc = hclust(dist(countries[, -1]))
> print(fit_hc)
Call:
hclust(d = dist(countries[, -1]))
Cluster method : complete
Distance : euclidean
Number of objects: 68
> plot(fit_hc)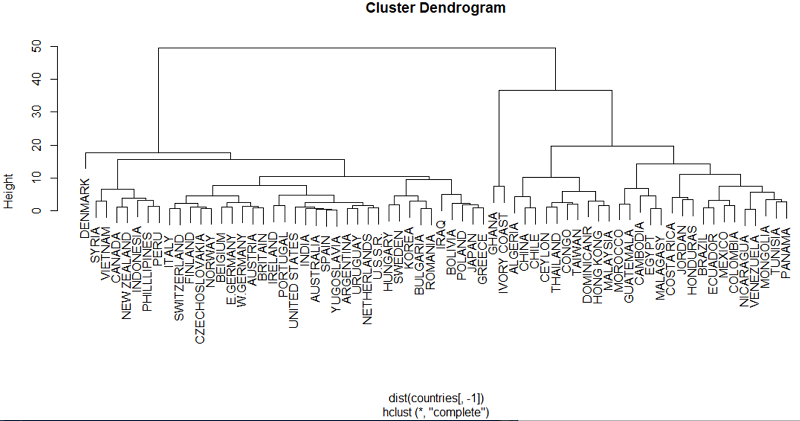
从系谱图可以看出,在图的最下端每个国家各占一个分支自成一类,越往上看,一条分支下的国家数越多,直至最上端所有国家聚为一类。在图的左侧以高度指标(Height)衡量树形图的高度,这一指标在下面将要提到的剪枝过程中将会用到。
接下来利用剪枝函数cutree()中的参数k和参数h分别控制输出10类别和Height=15时的系谱聚类结果:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31#利用剪枝函数cutree()中的参数k控制输出10类别的系谱聚类结果
> group_k10 = cutree(fit_hc, k = 10)
> group_k10
ALGERIA CONGO EGYPT GHANA IVORY COAST
1 2 3 4 4
……
YUGOSLAVIA AUSTRALIA NEW ZEALAND
6 6 7
> table(group_k10)
group_k10
1 2 3 4 5 6 7 8 9 10
3 7 5 2 9 21 7 10 3 1
#查看如上k=10的聚类结果中各类别样本
> sapply(unique(group_k10), function(g)countries$country[group_k10 == g])
[[1]]
[1] ALGERIA CHINA CHILE
68 Levels: ALGERIA ARGENTINA AUSTRALIA AUSTRIA BElGIUM BOLIVIA ... YUGOSLAVIA
……
[[10]]
[1] DENMARK
68 Levels: ALGERIA ARGENTINA AUSTRALIA AUSTRIA BElGIUM BOLIVIA ... YUGOSLAVIA
#利用剪枝函数cutree()中的参数h控制输出Height=15时的系谱聚类结果
> group_h15 = cutree(fit_hc, h = 15)
> table(group_h15)
group_h15
1 2 3 4 5 6
10 17 2 31 7 1
最后使用rect.hclus()函数从系谱图中选择查看聚类结果:1
2
3> plot(fit_hc)
> rect.hclust(fit_hc, k = 4, border = 'blue')
> rect.hclust(fit_hc, k = 7, border = 'red')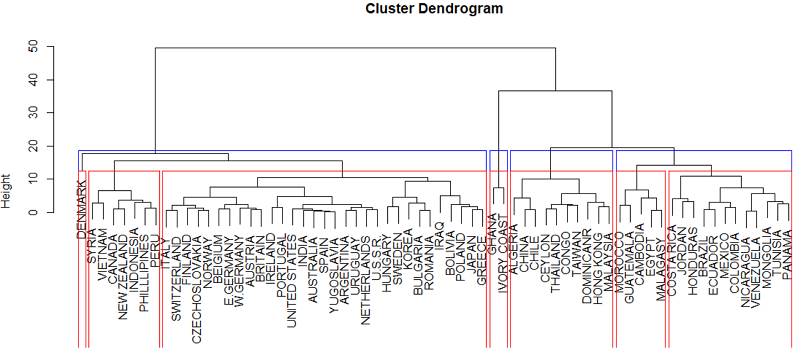
图中蓝色方框为4分类的结果,红色方框为7分类的结果。此图清晰地呈现了7类别是在原有的4类别的第一分类和第四分类的基础上进行再分类形成的。
2.4 密度聚类
基于密度的聚类算法相对于如上算法,其优势在于弥补了它们只能发现”类圆形”聚类簇的缺陷,该类算法由于是基于”密度”来聚类的,可以在具有噪声的空间数据库中发现任意形状的簇。DBSCAN叫算法是基于密度的聚类方法(Density-based Methods)中最常用的代表算法之一,具体算法可以参考Python部分笔记。
调用fpc包的dbscan()函数实现:dbscan (data, eps, Minpts = 5, scale = FALSE, method = c ("hybrid", "raw", "dist ") , seeds = TRUE, showplot = FALSE, countmode = NULL)
dbscan算法将数据点分为三类:
-核心点:在半径eps内含有超过MinPts数目的点。
-边界点:在半径eps内点的数量小于MinPts,但是落在核心点的邻域内的点。
-噪音点:既不是核心点也不是边界点的点。
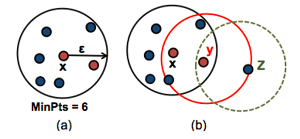
如上图(a),数据集中任意点x邻居(MinPts=6)都被标记为核心点,eps为半径。图(b)中,x为核心点,y的邻居小于MinPts(4<6),但它属于核心点x的最邻居,是边界点。z点既不是核心也不是边界点,它被称为噪声点或异常值。
针对该案例,首先查看大多数样本间的距离是在怎样一个范围,再以此距离作为半径参数的取值:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49> library(fpc)
> d = dist(countries[, -1])
> max(d);min(d)
[1] 49.56259
[1] 0.2236068
#加载ggplot2软件包,使用数据分段函数cut_interval()函数对各样本间的距离进行分段处理
> library(ggplot2)
#结合最大位最小值相差50左右,取居中段数为30
> interval = cut_interval(d, 30)
> table(interval)
interval
[0.224,1.87] (1.87,3.51] (3.51,5.16] (5.16,6.8] (6.8,8.45]
78 156 222 201 151
(8.45,10.1] (10.1,11.7] (11.7,13.4] (13.4,15] (15,16.7]
121 141 100 93 104
(16.7,18.3] (18.3,20] (20,21.6] (21.6,23.2] (23.2,24.9]
104 89 101 97 101
(24.9,26.5] (26.5,28.2] (28.2,29.8] (29.8,31.5] (31.5,33.1]
100 83 75 38 30
(33.1,34.8] (34.8,36.4] (36.4,38.1] (38.1,39.7] (39.7,41.3]
12 8 8 12 11
(41.3,43] (43,44.6] (44.6,46.3] (46.3,47.9] (47.9,49.6]
14 13 8 5 2
#找出所含样本点最多的区间
> which.max(table(interval))
(3.51,5.16]
3
#对半径取3、4、5,密度阈值为1至10,作双层循环
> for (i in 3:5) {
+ for (j in 1:10) {
+ ds = dbscan(countries[, -1], eps = i, MinPts = j)
+ print(ds)
+ }
+ }
dbscan Pts=68 MinPts=1 eps=3
1 2 3 4 5 6 7 8 9 10 11
seed 1 21 1 1 2 1 37 1 1 1 1
total 1 21 1 1 2 1 37 1 1 1 1
……
dbscan Pts=68 MinPts=9 eps=5
0 1 2
border 6 16 3
seed 0 9 34
total 6 25 37
dbscan Pts=68 MinPts=10 eps=5
0 1 2
border 7 18 4
seed 0 6 33
total 7 24 37
以上结果中,seed所对应的行表示的是相互密度可达的核心点,border所对应的行表示的是边缘点,0所对应列为噪音点的个数,列数1、2、3表示聚类结果的类别数,如最后一个结果总共聚类为2类,两类的样本个数(包含核心点和边界点)分别为24和37,噪音点个数为7。
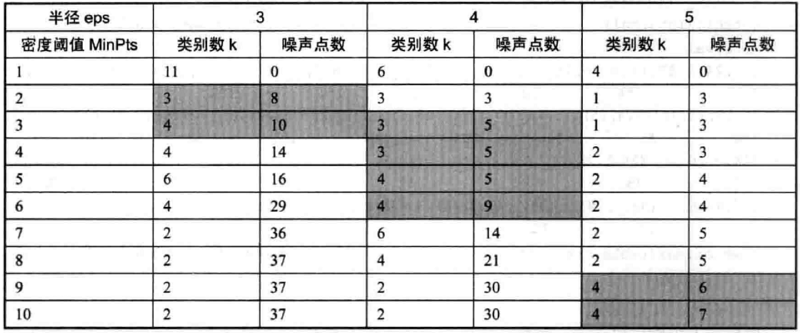
将30次的聚类结果整理汇总如上表,选择合适的参数值。一般来说,类别数应至少高于2类,否则进行聚类的意义不大;并且噪声点不应太多,若太多则说明参数条件过紧,参与有效聚类的样本点太少。符合条件的为阴影部分的取值。
选取其中三组参数(eps=3,MinPts=2;eps=4,MinPts=5;eps=5,MinPts=9)所对应的聚类结果进行绘图:1
2
3
4
5
6
7> ds5 = dbscan(countries[, -1], eps = 3, MinPts = 2)
> ds6 = dbscan(countries[, -1], eps = 4, MinPts = 5)
> ds7 = dbscan(countries[, -1], eps = 5, MinPts = 9)
> par(mfcol = c(1, 3))
> plot(ds5, countries[, -1], main = '5: MinPts=2 eps=3')
> plot(ds6, countries[, -1], main = '6: MinPts=5 eps=4')
> plot(ds7, countries[, -1], main = '7: MinPts=9 eps=5')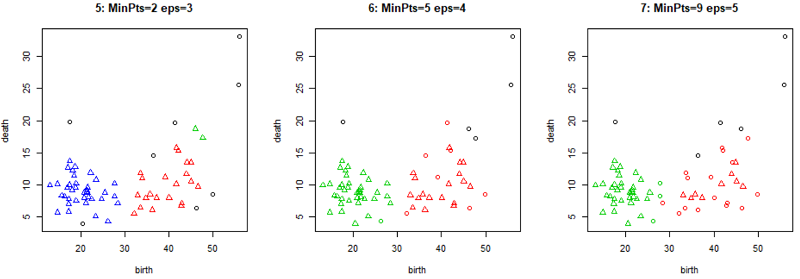
由以上过程,总结出DBSCAN算法参数取值的规律: 半径参数与阈值参数的取值差距越大,所得类别总数越小;半径参数相对于阈值参数较小时,越多的样本被判定为噪音点(这个规律其实不是特别准…)
2.5 期望最大化聚类
在使用该算法进行聚类时,它将数据集看作一个含有隐性变量的概率模型,并以实现模型最优化,即获取与数据本身性质最契合的聚类方式为目的,通过”反复估计”模型参数找出最优解,同时给出相应的最优类别数k。而”反复估计”的过程即是EM 算法的精华所在,这一过程由E-step (Expectation)和M-step(Maximization) 这两个步骤交替进行来实现。
调用mclust包的Mclust()、mclustBIC()、mclust2Dplot()、densityMclust()函数实现:Mclust(data, G = NULL, modelNames = NULL, prior = NULL, control = emControl(), initialization = NULL, warn = FALSE, ... )
mclustBIC()函数的参数设置与Mclust()基本一致,用于获取数据集所对应的参数化高斯混合模型的BIC值,而BIC值的作用即是评价模型的优劣,BIC值越高模型越优。mclust2Dplot()可根据EM算法所生成参数对二维数据制图。densityMclust()函数利用Mclust()的聚类结果对数据集中的每个样本点进行密度估计。
用Mclust()函数直接对数据进行期望最大化聚类:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15> library(mclust)
> fit_EM = Mclust(countries[, -1])
> summary(fit_EM)
----------------------------------------------------
Gaussian finite mixture model fitted by EM algorithm
----------------------------------------------------
Mclust EII (spherical, equal volume) model with 4 components:
log-likelihood n df BIC ICL
-418.415 68 12 -887.464 -893.5937
Clustering table:
1 2 3 4
2 13 17 36
以上结果显示,根据BIC选择出的最佳模型类型为EII,最优类别数为4,各类别分别含有2、13、17、36个样本。
对Mclust的聚类结果直接绘图:1
2
3
4
5
6
7> plot(fit_EM)
Model-based clustering plots:
1: BIC
2: classification
3: uncertainty
4: density
得到4张图形,分别为BIC图、分类图(classification)、概率图(uncertainty)以及密度图(density)。
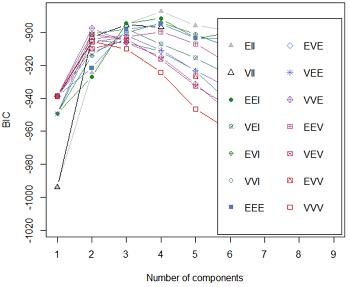
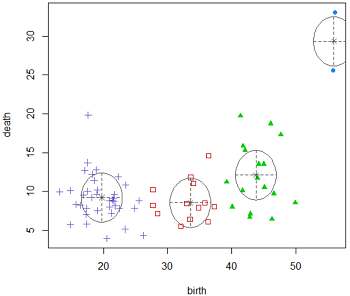
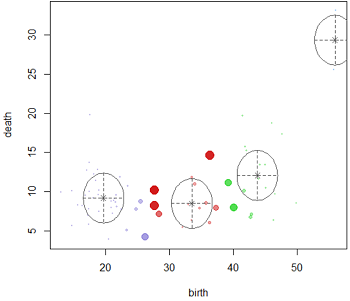
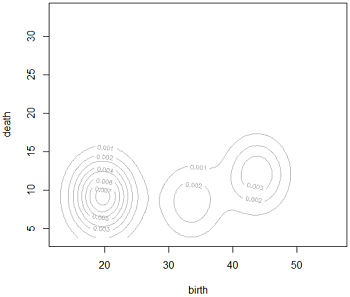
比如概率图不仅将各类别样本的主要分布区域用椭圆圈出,并标出了类别中心点,且以样本点图形的大小来显示该样本归属相应类别的概率大小。
下面用mclustBIC()函数来试试其他结果:1
2
3
4
5
6
7
8
9
10
11
12> countries_BIC = mclustBIC(countries[, -1])
> countries_BICsum = summary(countries_BIC, data = countries[, -1])
> countries_BICsum
Best BIC values:
EII,4 EEI,4 EVI,4
BIC -887.464 -891.670811 -894.177259
BIC diff 0.000 -4.206773 -6.713222
Classification table for model (EII,4):
1 2 3 4
2 13 17 36
得到的结果都是4分类的EII、EEI和EVI模型,其中EII模型的结果与Mclust()得到的结果一致。
也可以进一步对该结果进行绘图,这里就不放上来了。
3 思考
5种聚类算法内容很丰富,但是除了会调用软件包和使用函数,其实对有些算法的具体实现过程还是比较模糊的,希望以后有机会接触进一步学习。
这里也有个疑问:为什么同一批数据集,用k-means聚类得到是分为10类比较好,而用密度聚类和期望最大化聚类则是分为3或4类呢?以及如何评估聚类结果的好坏呢?
- 本笔记基于黄文和王正林编著的《数据挖掘:R语言实战》整理

